Dortmund, 11. November 2022
Facebook-Gründer Mark Zuckerberg und seine Ehefrau Priscilla Chan unterstützen zwei Forschungsvorhaben am ISAS mit 40.000 US-Dollar. Ein Jahr lang fördert die Chan Zuckerberg Initiative (CZI) die Entwicklung der Dortmunder Software für die Bildanalyse-Plattform napari: Am ISAS entwickeln die Forschungsgruppen AMBIOM* – Analysis of Microscopic BIOMedical Images sowie Spatial Metabolomics* künftig neue Plug-ins. Dadurch können Wissenschaftler:innen weltweit mikroskopische und chemische Aufnahmen besser analysieren – und dies kostenfrei.
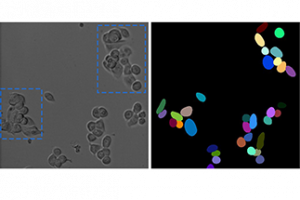
Die linke Abbildung zeigt eine Mikroskop-Aufnahme von Tumor-Zellen. Auf der rechten Seite ist die Segmentierung mittels gängiger Computerprogramme zu sehen. Sobald die Zellen dicht nebeneinander liegen oder überlappen (s. blaue Markierung) verschlechtert sich die Segmentierung. Das vollautomatische Tracking führt daher im Ergebnis zu Ungenauigkeiten.
© Prof. Dr. Barbara Grüner / Universitätsklinikum Essen
Zellbewegungen liefern in der Biomedizin wichtige Erkenntnisse, beispielsweise zur Entstehung verschiedener Krebserkrankungen. Um herauszufinden, wie sich Tumorzellen bei unterschiedlichen Krebsarten im Körper ausbreiten, untersuchen Forschende unter anderem deren Bewegung unter dem Mikroskop. Die dabei entstehenden Zeitraffer-Aufnahmen liefern zum Beispiel Aufschluss über die Beweglichkeit (Motilität) der Zellen. Für diese quantitative Analyse müssen Zellen, die überlappen, vorher voneinander getrennt werden (Segmentierung). Anschließend lassen sich ihre Wege verfolgen (Tracking).
Künstliche Intelligenz verbessert Zell-Tracking
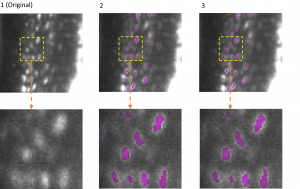
Die Bildanalyse hat sich in den vergangenen Jahren stark verbessert. Trotzdem kommt die Technik bei der Zellverfolgung aktuell noch an ihre Grenzen. „Zurzeit gibt es viele Mikroskopie-Szenarien, in denen eine automatisierte Analyse keine zufriedenstellenden Ergebnisse liefert – und es händischer Anpassung bedarf. Die Aufnahmen von 50 Zellen erzeugen große Datenmengen, die Forschende manuell kaum auswerten können“, erläutert Dr. Jianxu Chen, Experte für Künstliche Intelligenz (KI) und Leiter von AMBIOM. Daher möchte der Computerwissenschaftler mit seinem Team eine spezielle Software für das napari-System entwickeln. Diese soll Biomediziner:innen ermöglichen, unmittelbar in die automatische Auswertung einzugreifen und Fehler, beispielsweise bei der Segmentierung oder beim Tracking, zu beheben.
Die mit 20.000 US-Dollar geförderte Software „Human-in-the-loop Cell Tracking“ wird insgesamt drei Module (Segmentierung, Tracking und Analyse) beinhalten. Ihr Ziel unter Einsatz von KI: zum einen das Ergebnis der napari-Bildanalyse verbessern. Und zum anderen soll der Algorithmus mit den von Menschenhand eingebrachten Informationen mittels maschinellem Lernen trainiert werden. Dafür wird das AMBIOM-Team mit Immunologen am ISAS, dem Universitätsklinikum Essen und der Universität Duisburg-Essen kooperieren.
Massenspektrometrie-Bilddaten für den weltweiten Austausch
Auch an der Programmierung der zweiten, mit weiteren 20.000 US-Dollar geförderten, Software für biochemische Bildgebung ist AMBIOM beteiligt. Bei diesem Projekt handelt es sich um das erste napari-Plug-in für die Auswertung und Annotation von Massenspektrometrie-Bilddaten. Mit einem Massenspektrometer lassen sich in einer Probe Substanzen wie Metabolite (Stoffwechselprodukte) anhand ihrer Massen identifizieren. Mithilfe der bildgebenden Massenspektrometrie (Mass Spectrometry Imaging, MSI) können Wissenschaflter:innen beispielsweise Tumorgewebe auf metabolische Unterschiede bei subzellulärer Auflösung untersuchen. Auf diese Weise gewinnen sie Informationen über die räumliche Verteilung der Moleküle und können diese mit morphologischen Auffälligkeiten im Gewebe vergleichen.
Dr. Prasad Phapale, Chemiker und Leiter von Spatial Metabolomics, strebt eine bessere Integration (Multiplexing) der MSI-Daten mit anderen Bildformaten an. So soll das Plug-in „Biochemical Annotations of Mass Spectrometry Imaging Data“ für napari die biochemische Annotation von MSI-Daten mit Bild-Koregistrierung (Bildfusion) erlauben. Kurzum: Es soll Wissenschaftler:innen weltweit ermöglichen, ihre MSI-Ergebnisse mit Metaboliten-Datenbanken abzugleichen sowie die räumlichen Informationen aus ihrer Aufnahme mit denen ergänzender Analysemethoden, etwa der Mikroskopie, abzugleichen. Innerhalb der Forschenden-Gemeinschaft ließe sich das Wissen so besser teilen.
Über napari
napari ist ein Open-Source-Tool, das eine leistungsstarke Visualisierung mehrdimensionaler Bilder, beispielsweise aus der Mikroskopie, ermöglicht. napari basiert auf der Programmiersprache Python. An der Plattform beteiligt sich eine weltweit wachsenden Community von Forschenden und Software-Entwickler:innen. Die CZI fördert napari über ihr Imaging-Programm mit dem Ziel, Biolog:innen den Zugang zu neuen Methoden der Bildanalyse auf Basis des maschinellen Lernens zu erleichtern.
* Das Bundesministerium für Bildung und Forschung (BMBF) fördert die MSCoreSys-assoziierte Nachwuchgruppe AMBIOM – Analysis of Microscopic BIOMedical Images (Förderkennzeichen 161L0272). Das BMBF fördert ebenfalls die MSCoreSys-assoziierte Nachwuchsgruppe Spatial Metabolomics (Förderkennzeichen 161L0271).