Dortmund, 24. Januar 2024
Künstliche Intelligenz (KI) hat sich mittlerweile als unverzichtbare Komponente der Auswertung mikroskopischer Daten etabliert. Doch während die KI-Modelle immer besser und komplexer werden, steigt auch der Rechenaufwand – und damit einhergehend der Energieverbrauch. Forschende des Leibniz-Instituts für Analytische Wissenschaften (ISAS) und der Universität Peking haben deswegen eine kostenfreie Kompressionssoftware entwickelt, mit der Wissenschaftler:innen bereits bestehende Bioimaging-KI-Modelle schneller und mit wesentlich geringerem Energieaufwand ausführen können. Ihre nutzerfreundliche Toolbox namens EfficientBioAI (Open Source) haben die Forschenden nun im Fachjournal Nature Methods vorgestellt.
Moderne Mikroskopieverfahren produzieren eine Vielzahl hochauflösender Bilder, einzelne Datensätze können tausende davon umfassen. Um die Datenmengen zuverlässig analysieren zu können, nutzen Wissenschaftler:innen häufig KI-gestützte Software. Mit immer komplexeren KI-Modellen kann sich die Latenzzeit (Verarbeitungszeit) für Bilder jedoch deutlich erhöhen. „Eine hohe Netzwerklatenz, beispielsweise bei besonders großen Bildern, führt zu einer höheren Rechenleistung und schließlich zu einem gesteigerten Energieverbrauch“, sagt Dr. Jianxu Chen, Leiter der Nachwuchsgruppe AMBIOM – Analysis of Microscopic BIOMedical Images am ISAS.

Unsere Versuche haben gezeigt, dass EfficientBioAI die Effizienz neuronaler Netzwerke bei Bioimaging-Analysen deutlich erhöhen kann, ohne die Genauigkeit der Modelle einzuschränken.
Dr. Jianxu Chen
Neue Anwendungsgebiete für eine bekannte Technik
Um hohe Latenzen bei der Bildanalyse zu vermeiden, insbesondere bei Geräten mit begrenzter Rechenleistung, verwenden die Forschenden komplexe Algorithmen und komprimieren damit die KI-Modelle. Das heißt, sie reduzieren die Menge der Berechnungen in den Modellen, während diese eine vergleichbare Vorhersagegenauigkeit beibehalten. „Modellkomprimierung ist eine Technik, die bei der digitalen Bildverarbeitung, der sogenannten Computer Vision, und der KI weit verbreitet ist, um Modelle leichter und grüner zu machen“, erklärt Chen. Dabei kombinieren Forschende verschiedene Strategien, um den Speicherverbrauch zu reduzieren, die Modellinferenz, also den „Denkprozess“ des Modells zu beschleunigen – und somit Energie zu sparen. Zum Einsatz kommt beispielsweise Pruning, das überflüssige Knoten aus dem neuronalen Netzwerk entfernt. „In der Bioimaging-Gemeinschaft sind diese Techniken häufig unbekannt. Daher wollten wir eine gebrauchsfertige und einfache Lösung entwickeln, um sie auf gängige KI-Tools beim Bioimaging anzuwenden“, sagt Yu Zhou, Erstautor der Publikation und Doktorand bei AMBIOM.

Yu Zhou ist seit September 2022 Doktorand in der Nachwuchsgruppe AMBIOM – Analysis of Microscopic BIOMedical Images am ISAS. Zuvor hat er Biomedizintechnik in Schweden, China und in der Schweiz studiert.
© ISAS
Bis zu ca. 81 Prozent Energieeinsparung
Um ihre neue Toolbox auf die Probe zu stellen, haben die Forschenden um Chen ihre Software an mehreren realen Anwendungen getestet. Bei unterschiedlicher Hardware und verschiedenen Bioimaging-Analyseaufgaben konnten die Komprimierungstechniken die Latenzzeit erheblich verringern und den Energieverbrauch zwischen 12,5 bis 80,6 Prozent senken. „Unsere Versuche haben gezeigt, dass EfficientBioAI die Effizienz neuronaler Netzwerke bei Bioimaging-Analysen deutlich erhöhen kann, ohne die Genauigkeit der Modelle einzuschränken“, resümiert Chen. Die Energieeinsparungen verdeutlicht er am Beispiel des allgemein verwendeten Cellpose-Modells: Würden eintausend Nutzer:innen die Toolbox nutzen, um das Modell zu komprimieren und auf den Jump-Target-ORF-Datensatz (etwa eine Million Mikroskopbilder von Zellen) anwenden, könnten sie Energie einsparen, die circa den Emissionen einer Autofahrt von etwa 7.300 Meilen (ca. 11.750 Kilometer) entsprechen.
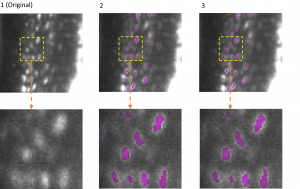
Die Anpassungsfähigkeit von EfficientBioAI wurde anhand mehrerer Anwendungen getestet. Ein Beispiel ist die semantische Segmentierung bei 3D-Aufnahmen. Dabei identifiziert das KI-Model einzelne spezifische Strukturen innerhalb eines dreidimensionalen zellulären Umfelds. Die Abbildung zeigt die Segmentierung von Osteozyten (reife Knochenzellen) in Aufnahmen von Mäuseknochen. Diese wurden mit dem Lichtblatt-Fluoreszenzmikroskop aufgenommen.Die Darstellung zeigt die Originalaufnahme (Spalte 1), die Segmentierung durch das Modell FNet 3D (Spalte 2) aus der MMV_Im2Im Toolbox für die Bild-zu-Bild-Transformation, und die Segmentierung nach der Kompression von FNet 3D durch EfficientBioAI (Spalte 3). Der Vergleich (Spalten 2 und 3) verdeutlicht: Die Kompression beeinflusst keineswegs die Genauigkeit der Segmentierung.
© ISAS / Prof. Dr. Anika Grüneboom (Bioimaging)
Keine speziellen Kenntnisse notwendig
Die Autor:innen legen viel Wert darauf, EfficientBioAI möglichst vielen Wissenschaftler:innen in der biomedizinischen Forschung zugänglich zu machen. Forschende können die Software installieren und nahtlos in bereits existierende PyTorch-Bibliotheken (Open-Source-Programmbibliothek für die Programmiersprache Python) integrieren. Für einige sehr verbreitete Modelle wie etwa Cellpose, können Forschende die Software nutzen, ohne selbst etwas am Code ändern zu müssen. Für spezifische Änderungswünsche stellt die Gruppe außerdem mehrere Demos und Tutorials zur Verfügung. Mit nur ein paar geänderten Code-Zeilen lässt sich die Toolbox dann auch für angepasste KI-Modelle verwenden.
Über EfficientBioAI
EfficientBioAI ist eine gebrauchsfertige und frei zugängliche Kompressionssoftware für KI-Modelle im Bioimaging. Die „plug-and-play“-Toolbox ist zwar für den Standardgebrauch einfach gehalten, bietet aber anpassbare Funktionen. Dazu gehören etwa adaptierbare Kompressionsstufen sowie müheloses Umschalten zwischen Hauptprozessor (Central Processing Unit, CPU) und Grafikprozessor (Graphics Processing Unit, GPU). Die Forschenden in Dortmund und Peking arbeiten bereits daran, die Software neben Linux (Ubuntu 20.04, Debian 10) und Windows 10 auch für MacOS verfügbar zu machen. Aktuell liegt der Fokus der Toolbox darauf, die Inferenzeffizienz bei vortrainierten Modellen zu verbessern, anstatt die Effizienz während der Trainingsphase zu steigern. Die Forschenden entwickeln die Toolbox jedoch kontinuierlich weiter.
EfficientBioAI ist bei GitHub verfügbar unter https://github.com/MMV-Lab/EfficientBioAI.
(Wir möchten darauf hinweisen, dass nach Aktivierung des Links Daten an GitHub übermittelt werden.)
Originalpublikation
https://www.nature.com/articles/s41592-024-02167-z
Das Bundesministerium für Bildung und Forschung (BMBF) fördert die MSCoreSys-assoziierte Nachwuchsgruppe AMBIOM – Analysis of Microscopic BIOMedical Images unter dem Förderkennzeichen 161L0272.
Downloads
-
PDF (0.23MB)
Pressemitteilung: EfficientBioAI: Neue Open-Source-Software macht KI-Modelle leichter & grüner
-
JPG (0.17MB)
Porträt Dr. Jianxu Chen
Das Foto steht für die redaktionelle Nutzung ausschließlich bei der Berichterstattung zu EfficientBioAI zur Verfügung.
-
PNG (1.3MB)
Porträt Yu Zhou
Das Foto steht für die redaktionelle Nutzung ausschließlich bei der Berichterstattung zu EfficientBioAI zur Verfügung.
-
PNG (0.45MB)
Abbildung Semantische 3D-Segmentierung
Das Foto steht für die redaktionelle Nutzung ausschließlich bei der Berichterstattung zu EfficientBioAI zur Verfügung.